Kapitel 11 Data Munging
Ah ja, die wunderbare Welt des Data Munging – dem Dammelbegriff für “Zeug mit Daten machen damit wir da besser mit arbeiten können oder so”. Hierzu gehören so spaßige Themen wie “recoding”, “reformatting”, “restructuring”, “labelling”, “transforming”, “reshaping” und viele andere lustige Begriffe die teilweise kongruent sind, und auch sonst ist das eher so ein Bereich á la learning by doing.
Wie schon beim Datenimport erwähnt gibt es in der Regel kein Patentrezept zru Datenbereinigung, aber es gibt gängige Anwendungsfälle, und dementsprechend auch populäre Lösungswege für selbige Fälle.
In diesem Kapitel widmen wir uns also einigen dieser gängigen Aufgaben und probieren das Ganze anhand unserer Beispieldatensätze aus.
11.1 Vorab: Pipes!
Lasst mich euch euer neues Stück Lieblingsyntax vorstellen: %>%.
Das ist die pipe, im Code gesprochen als “dann”. Nichts verbessert die Lesbarkeit und Nachvollziehbarkeit von Code so nachhaltig wie großzügige Verwendungen dieses kleinen Operators.
Die pipe steht zwischen zwei Funktionen, und setzt das linke Element als erstes Argument in die rechte Funktion. Sprich: f(x) %>% g() ist äquivalent zu g(f(x)).
Ich sehe schon, wir brauchen Beispiele:
Mal angenommen wir haben einen Vektor von Zahlen, und wollen diese zuerst quadrieren, dann aufsummieren, dann die Wurzel aus dem Ergebnis ziehen und dann auf zwei Nachkommastellen runden. Wieso sollten wir das tun? Für Übungszwecke. Alles andere wäre ja albern.
#> [1] 19.16Das sieht ziemlich unübersichtlich aus, oder? Wir müssen den Code praktisch von x angefangen von innen nach außen lesen, um zu verstehen, was da eigentlich passiert.
Eine Möglichkeit das zu umgehen, wäre die Erstellung von Zwischenergebnissen:
Das ist… möglich, aber auch das wird irgendwann unübersichtlich, und solange ihr nicht jedem Zwischenergebnis einen anderen Namen gebt, wird das auch irgendwann schwer nachvollziehbar, insbesondere wenn ihr einen Fehler in eurem Code habt und Zwischenergebnisse nachvollziehen wollt.
Mit der pipe sähe das dann so aus:
#> [1] 19.16Das sieht jetzt erstmal noch nicht so besonders nach Verbesserung aus, aber achtet darauf, wie wir den Prozess jetzt ganz einfach von links nach rechts lesen können, oder mit mehrzeiliger Formatierung:
#> [1] 19.16Pipelines in dieser Art werdet ihr noch sehr viele sehen, und früher oder später werdet ihr sie zu schätzen lernen, just trust me on this one.
Ein komplexeres Beispiel aus einem meiner alten Projekte sieht etwa so aus:
library(rvest)
library(dplyr)
library(stringr)
happiness <- read_html("https://en.wikipedia.org/wiki/World_Happiness_Report") %>%
html_table(fill = TRUE, trim = TRUE) %>%
extract2(1) %>%
select(Country, Score) %>%
mutate(Country = str_trim(Country, "both")) %>%
set_colnames(c("country", "happiness_score"))Was da passiert ist etwas Folgendes:
- Wir erstellen ein Objekt
happiness, dann… - lesen eine Wikipedia-Seite ein via
read_html, dann… - holen wir da Tabellen raus mit
html_table, dann… - extrahieren wir das erste Element via
extract2, dann… - wählen wir via
selectzwei Spalten der Tabelle aus, dann… - wenden wir
str_trimauf eine Variable an inmutate, dann… - setzen wir die Variablennamen via
set_colnames.
Fertig!
Und das alles in nur einer Pipeline.
Ihr müsst den Code oben nicht inhaltlich nachvollziehen können, aber ihr seht vermutlich, dass die Struktur deutlich einfacher zu verstehen ist, als eine lange Verschachtelung mehrer Funktionen oder eine Reihe von Befehlen mit mehreren Zwischenschritten.
Und das ist die Stärke der pipe, und das ist das zentrale Prinzip in allen tidyverse-packages.
11.1.1 magrittr-Boni
Der %>%-Operator kommt ja ursprünglich aus dem magrittr-package, und auch wenn viele packages wie dplyr, tidyr oder auch tadaatoolbox den Operator auch mitbringen, hat pures magrittr noch einige nette Boni für Pipe-Konstruktionen
library(magrittr)
c(1, 4, 7, 4, 8, 19, 33, 42, 12, 4, 16) %>%
divide_by(5) %>%
add(9) %>%
raise_to_power(2)#> [1] 84.64 96.04 108.16 96.04 112.36 163.84 243.36 302.76 129.96 96.04
#> [11] 148.84Die Funktionen add, divide_by und raise_to_power sind nur andere Versionen der Rechenoperatoren mit entsprechenden Namen, +, / und ^, die sich besser für Pipelines eignen.
11.2 Variablen verändern oder anlegen
Als Beispiel lesen wir mal den gotdeaths_books-Datensatz ein:
#> Parsed with column specification:
#> cols(
#> Name = col_character(),
#> Allegiances = col_character(),
#> `Death Year` = col_double(),
#> `Book of Death` = col_double(),
#> `Death Chapter` = col_double(),
#> `Book Intro Chapter` = col_double(),
#> Gender = col_double(),
#> Nobility = col_double(),
#> GoT = col_double(),
#> CoK = col_double(),
#> SoS = col_double(),
#> FfC = col_double(),
#> DwD = col_double()
#> )Ihr seht, dass Spalten Namen haben wie Death Year, was wegen des Leerzeichens dazwischen etwas unhandlich ist. Zusätzlich haben wir Spalten Gender und Nobility, die mit 1 und 0 kodiert sind, die wären mit Labels lesbarer.
11.2.1 Spaltennamen
Spaltennamen lassen sich einfach mit names() anzeigen und ändern:
# Spaltennamen anzeigen
names(gotdeaths_books)
# Spaltennamen ändern
names(gotdeaths_books) <- c("Name", "Allegiances", "Death_Year", "Book_of_Death",
"Death_Chapter", "Book_Introduced", "Gender", "Nobility",
"GameOfThrones", "ClashOfKings", "SongOfStorms", "FeastForCrows",
"DanceWithDragons")…aber so müssen wir einen neuen Vektor mit der gleichen Anzahl an Elementen wie Spalten im Datensatz angeben, das ist ziemlich nervig, wenn wir nur einzelne Spalten umbenennen wollen.
Zum glück gibt’s da was von dplyr:
library(dplyr)
gotdeaths_books <- gotdeaths_books %>%
rename(Death_Year = 'Death Year',
Death_Chapter = 'Death Chapter',
Death_Book = 'Book of Death',
Book_Intro = 'Book Intro Chapter')So müssen wir nur die Variablen angeben, die wir umbenennen wollen. Der neue Variablennamen steht links in rename, dann rechts der Name der aktuellen Variable, in diesem Fall in ' ' wegen der Leerzeichen.
Wir speichern das Ganze auch gleich wieder via gotdeaths_books <- in das gleiche Objekt.
11.2.2 Rekodieren
Als nächsten wollen wir die Variablen Gender und Nobility rekodieren, damit die numerischen Werte durch was aussagekräftigeres ersetzt werden. Das können wir am einfachsten mit mutate aus dplyr machen:
gotdeaths_books <- gotdeaths_books %>%
mutate(Gender = factor(Gender, levels = c(0, 1), labels = c("Female", "Male")),
Nobility = factor(Nobility, levels = c(0, 1), labels = c("No", "Yes")))Die Funktion mutate funktioniert nach dem gleichen Schema wie rename: Links steht der Namen der Spalte die wir erstellen (in diesem Fall überschreiben) wollen, und rechts neben dem = steht ein Ausdruck, der eine Variable mit gleicher Anzahl an Elementen zurückgibt. In diesem Fall ist die Funktion factor, angewandt auf die jeweils zu rekodierende Variable.
factor() erstellt einen Vektor des Typs, well, factor, mit numerischen Werten (levels), praktisch den Merkmalsausprägungen, und mit character Labels (labels). Beide nennen wir in der Funktion explizit. Das Resultat ist, dass die Variable Gender immer noch die levels 0, 1 hat, aber jetzt zusätzlich die labels "Männlich", "Weiblich".
Nachdem wir den Befehl oben ausgeführt haben können wir uns die Variablen in der Konsole angucken:
#> [1] "factor"#> [1] "factor"#> # A tibble: 917 x 2
#> Gender Nobility
#> <fct> <fct>
#> 1 Male Yes
#> 2 Male Yes
#> 3 Male Yes
#> 4 Male Yes
#> 5 Male Yes
#> 6 Male Yes
#> 7 Male Yes
#> 8 Female Yes
#> 9 Male Yes
#> 10 Male No
#> # … with 907 more rowsDann ist da noch eine Sache mit den Allegiances. Wenn ihr euch die Variable anschaut, seht ihr, dass da manchmal “Stark” und manchmal “House Stark” etc. steht. Wenn wir jetzt aber nach der Zugehörigkeit gruppieren wollen in Tabellen und Plots, dann wären das ja Duplikate.
Das zu beheben ist leider etwas komplizierter, wenn wir’s sauber machen wollen, aber haltet durch.
library(stringr)
gotdeaths_books <- gotdeaths_books %>%
mutate(Allegiances = str_replace(Allegiances, pattern = "House\\ ", replacement = ""))
gotdeaths_books %>% count(Allegiances)#> # A tibble: 12 x 2
#> Allegiances n
#> <chr> <int>
#> 1 Arryn 30
#> 2 Baratheon 64
#> 3 Greyjoy 75
#> 4 Lannister 102
#> 5 Martell 37
#> 6 Night's Watch 116
#> 7 None 253
#> 8 Stark 108
#> 9 Targaryen 36
#> 10 Tully 30
#> 11 Tyrell 26
#> 12 Wildling 40Fixed it.
Okay, was ist da passiert?
- Anwendung von
mutateso wie eben. VariableAllegiancesersetzen durch eine modifizierte Version - Wir haben das package
stringrgeladen und benutzt für die Funktionstr_replace, darin…
2.1. Benutzen wir die VariableAllegiances…
2.2. Suchen das “Muster”"House\\ ", das steht für “Das wortHousemit einem Leerzeichen danach”
2.3. Ersetzen das gesuchte Muster durch"", also leeren Text
- Das Resultat ist die Variable
Allegiances, aber überall wurdeHouseentfernt
Das was wir hier gemacht haben fällt unter die Themen string manipulation und regular expressions.
Das müsst ihr nicht sofort verstehen oder jetzt recherschieren, aber es kann helfen das zu können. Kommt alles mit der Zeit und lässt sich prima googlen, weil das in vielen Bereichen häufig vorkommt.
11.2.3 Klassieren
Für diesen Anwendungsfall nehmen wir am besten wieder den qmsurvey-Datensatz, weil es bei den Game of Thrones-Daten so wenig zu klassieren gibt.
Zum klassieren (also Sonderfall des Rekodierens) haben wir mehrere Optionen in R.
Die erste ist aus car:
library(dplyr) # Für mutate und %>%
library(car) # Für recode
qmsurvey <- qmsurvey %>%
mutate(schlaf_k = recode(schlafstunden, "lo:7 = 1; 7.5:9 = 2; 9:hi = 3"))Leider benutzt sich recode etwas umständlich, aber der Befehl liest sich etwa so:
- “Alle Werte vom niedrigsten (
lo, sprich”low“) bis7sollen zu1werden” - “Alle Werte von
7.5bis9sollen zu2werden” - “Alle Werte von
9bis zum höchsten (hi, sprich”high“) sollen zu3werden”
Eine Alternative Möglichkeit aus sjmisc ist split_var:
Hier steht n = 3 für die Anzahl der Gruppen, die wir gerne hätten. Das Resultat hat in diesem Fall allerdings nur 2 Gruppen, vermutlich weil die Spannweite der Werte relativ klein ist.
Eine dritte Variante wäre dplyr mit case_when, und erfordert Logik:
qmsurvey <- qmsurvey %>%
mutate(schlaf_k = case_when(
schlafstunden >= 9 ~ 3,
schlafstunden < 9 ~ 2,
schlafstunden <= 7 ~ 1
))Das liest sich so:
- Alle Werte größer gleich
9sollen zu3werden - Alle Werte kleiner als
9sollen zu2werden - Alle Werte kleiner gleich
7sollen zu1werden
Benutzt eine diese Varianten, je nachdem welche ihr am Verständlichsten findet.
Und ja, es gibt noch viele andere Möglichkeiten, aber irgendwann ist auch mal gut.
11.3 Summary Statistics
#> # A tibble: 1 x 3
#> m_alter sd_alter median_alter
#> <dbl> <dbl> <dbl>
#> 1 21.7 3.82 20qmsurvey %>%
group_by(rauchen) %>%
summarize(m_alter = mean(alter),
sd_alter = sd(alter),
median_alter = median(alter))#> # A tibble: 3 x 4
#> rauchen m_alter sd_alter median_alter
#> <fct> <dbl> <dbl> <dbl>
#> 1 In Gesellschaft 22.6 3.55 22
#> 2 Ja 23 5.20 21
#> 3 Nein 21.4 3.73 2011.4 Der dplyr-Workflow
#> # A tibble: 12 x 2
#> Allegiances n
#> <chr> <int>
#> 1 Tyrell 2
#> 2 Martell 3
#> 3 Arryn 5
#> 4 Targaryen 9
#> 5 Tully 9
#> 6 Greyjoy 22
#> 7 Wildling 23
#> 8 Baratheon 24
#> 9 Lannister 30
#> 10 Stark 46
#> 11 Night's Watch 56
#> 12 None 76Als plot:
11.5 Format: Wide vs. Long
11.5.1 Beispiel 1
#> # A tibble: 917 x 5
#> GoT CoK SoS FfC DwD
#> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 1 1 1 1 0
#> 2 0 0 1 0 0
#> 3 0 0 0 0 1
#> 4 0 0 0 0 1
#> 5 0 0 1 0 0
#> 6 0 1 1 0 0
#> 7 1 0 1 1 0
#> 8 1 1 1 0 1
#> 9 0 1 0 1 0
#> 10 0 0 1 0 0
#> # … with 907 more rowslibrary(tidyr)
gotdeaths_books %>%
select(GoT, CoK, SoS, FfC, DwD) %>%
gather(key = Book, value = Appearance)#> # A tibble: 4,585 x 2
#> Book Appearance
#> <chr> <dbl>
#> 1 GoT 1
#> 2 GoT 0
#> 3 GoT 0
#> 4 GoT 0
#> 5 GoT 0
#> 6 GoT 0
#> 7 GoT 1
#> 8 GoT 1
#> 9 GoT 0
#> 10 GoT 0
#> # … with 4,575 more rowsgotdeaths_books %>%
select(GoT, CoK, SoS, FfC, DwD) %>%
gather(key = Book, value = Appearance) %>%
filter(Appearance > 0)#> # A tibble: 1,474 x 2
#> Book Appearance
#> <chr> <dbl>
#> 1 GoT 1
#> 2 GoT 1
#> 3 GoT 1
#> 4 GoT 1
#> 5 GoT 1
#> 6 GoT 1
#> 7 GoT 1
#> 8 GoT 1
#> 9 GoT 1
#> 10 GoT 1
#> # … with 1,464 more rowsgotdeaths_books %>%
select(GoT, CoK, SoS, FfC, DwD) %>%
gather(key = Book, value = Appearance) %>%
filter(Appearance > 0) %>%
group_by(Book) %>%
summarize(Character_Appearances = sum(Appearance))#> # A tibble: 5 x 2
#> Book Character_Appearances
#> <chr> <dbl>
#> 1 CoK 324
#> 2 DwD 261
#> 3 FfC 250
#> 4 GoT 250
#> 5 SoS 389library(ggplot2)
gotdeaths_books %>%
select(GoT, CoK, SoS, FfC, DwD) %>%
gather(key = Book, value = Appearance) %>%
filter(Appearance > 0) %>%
group_by(Book) %>%
summarize(Appearances = sum(Appearance)) %>%
ggplot(aes(x = reorder(Book, Appearances), y = Appearances)) +
geom_col(color = "black", alpha = .75) +
labs(title = "A Song of Ice and Fire",
subtitle = "Character Appearances per Book",
x = "Book", y = "Character Appearances")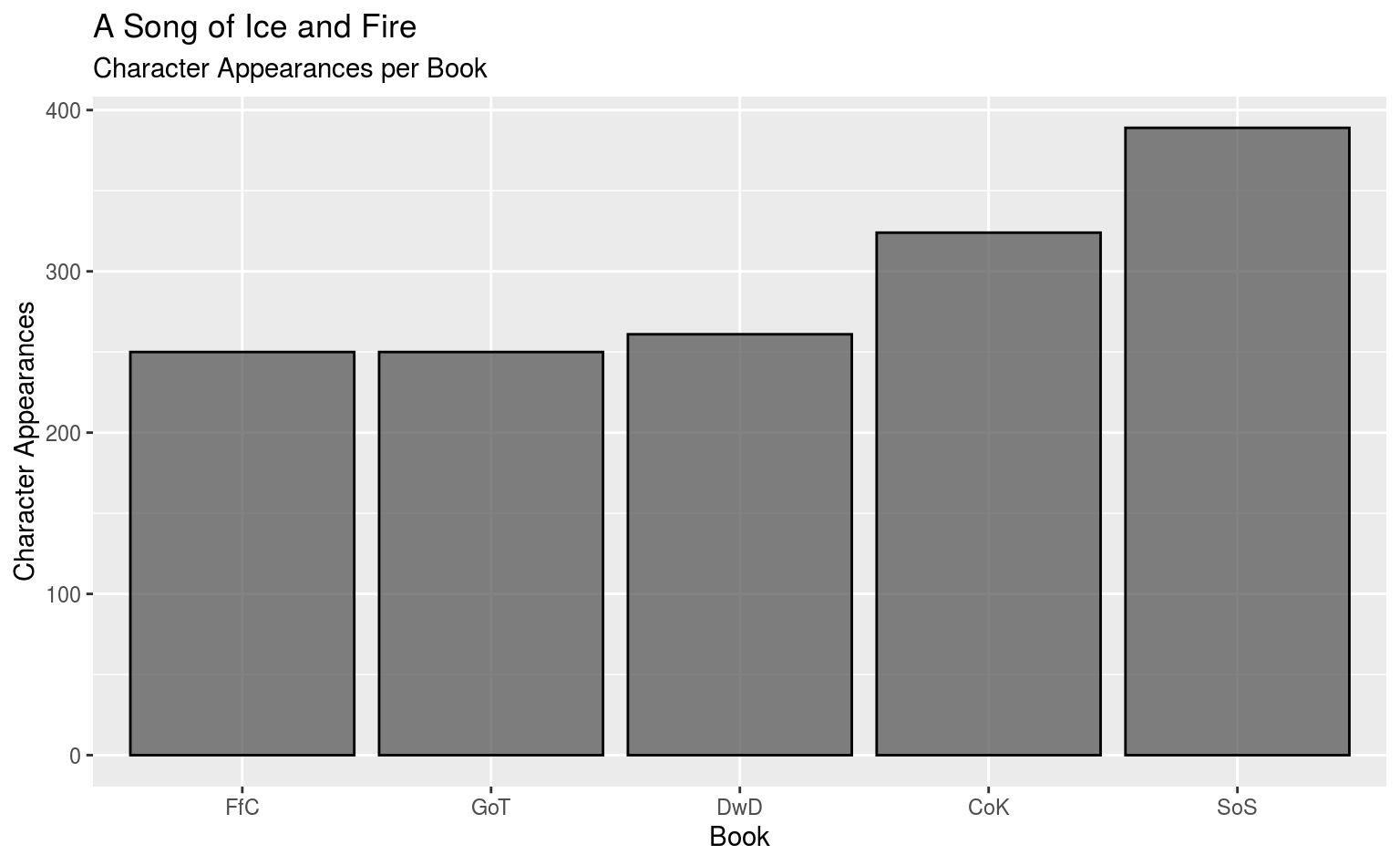
11.5.2 Beispiel 2
gotdeaths_books %>%
gather(key = Book, value = Appearance, GoT, CoK, SoS, FfC, DwD) %>%
select(Name, Book, Appearance)#> # A tibble: 4,585 x 3
#> Name Book Appearance
#> <chr> <chr> <dbl>
#> 1 Addam Marbrand GoT 1
#> 2 Aegon Frey (Jinglebell) GoT 0
#> 3 Aegon Targaryen GoT 0
#> 4 Adrack Humble GoT 0
#> 5 Aemon Costayne GoT 0
#> 6 Aemon Estermont GoT 0
#> 7 Aemon Targaryen (son of Maekar I) GoT 1
#> 8 Aenys Frey GoT 1
#> 9 Aeron Greyjoy GoT 0
#> 10 Aethan GoT 0
#> # … with 4,575 more rowsgotdeaths_books %>%
gather(key = Book, value = Appearance, GoT, CoK, SoS, FfC, DwD) %>%
filter(Appearance > 0) %>%
group_by(Name) %>%
summarize(Appearances = sum(Appearance)) %>%
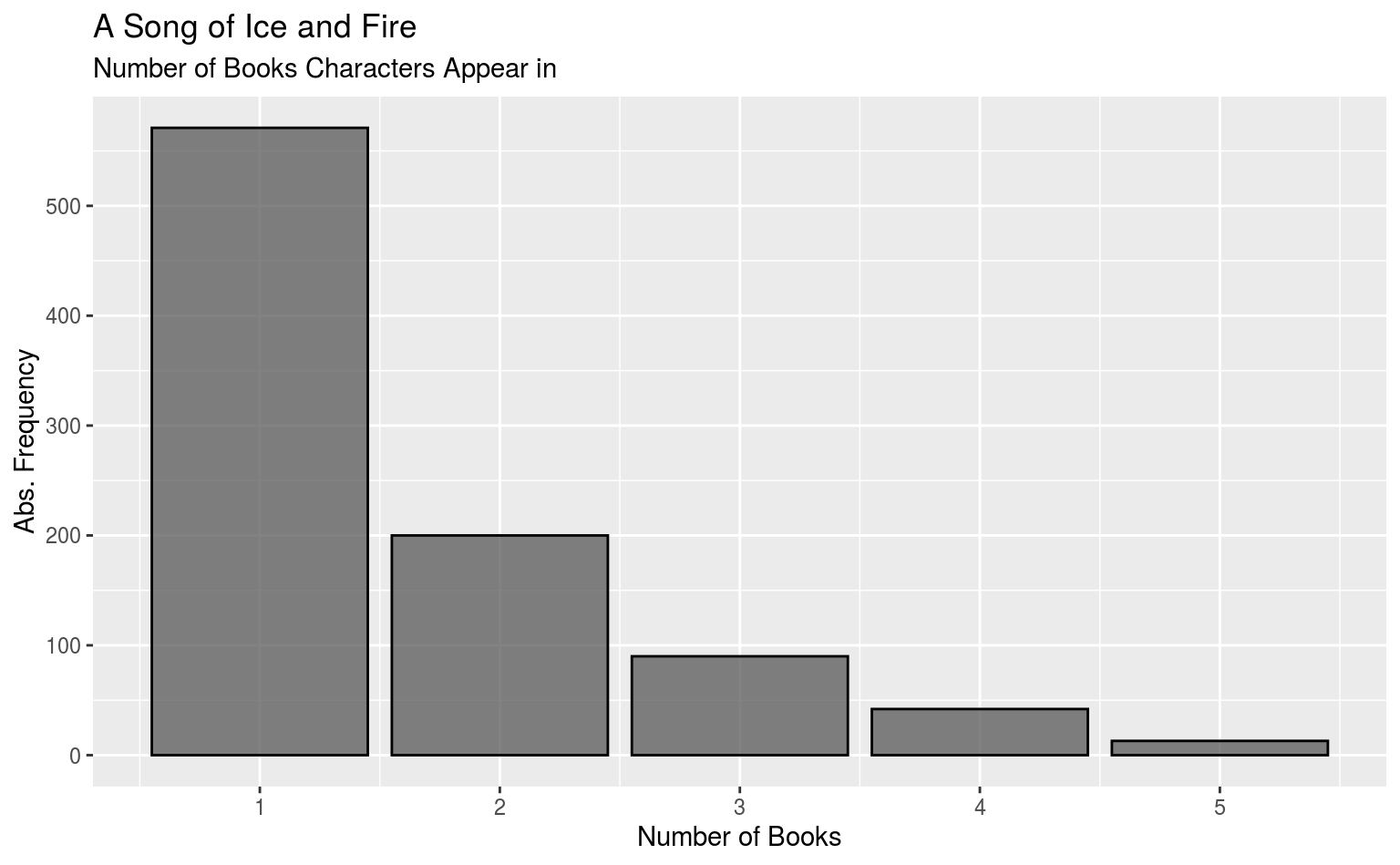
ggplot(aes(x = Appearances)) +
geom_bar(alpha = .75, color = "black") +
scale_y_continuous(breaks = seq(0, 1000, 100),
minor_breaks = seq(0, 1000, 50)) +
labs(title = "A Song of Ice and Fire",
subtitle = "Number of Books Characters Appear in",
x = "Number of Books", y = "Abs. Frequency")