Kapitel 6 Packages
Fast jede Software hat Erweiterungen irgendeiner Art. Manche haben extensions, andere plug-ins, wieder andere haben add-ons. Unterschiedliche Terminologie für dasselbe Prinzip: Mehr Features durch Erweiterungen anderer Leute.
Bei Programmiersprachen heißt sowas meistens library oder package11.
R hat sowas natürlich auch, als populäre open-source Software. Hier heißt sowas packages und besteht aus Funktionen, die andere Leute für bestimmte Anwendungsfälle geschrieben haben, und durch ein Verteilungssystem verfügbar machen, sodass wir alle sie benutzen können.
Der Kern von R wird auch base genannt und umfasst die wichtigstens Grundfunktionen — mit denen kommen wir auch schon relativ weit, wir können zum Beispiel problemlos diverse Statistiken berechnen und sogar Visualisierungen machen, aber wir wollen natürlich mehr, einfacher, schneller und besser.
6.1 Installieren, Laden, Updaten
Packages ladet ihr aus dem Internet runter, woraufhin sie ggf. kompiliert und in eure R-library eingepflegt werden müssen.
Das klingt kompliziert, und deshalb passiert das auch alles automatisch!
Nehmen wir als Beispiel-Package mal tadaatoolbox — ein Package von Lukas und Tobi, das insbesondere für einige Sachen aus QM2 geschrieben wurde, um Dinge etwas einfacher oder zumindest schöner zu machen.
Wir installieren das package mit einem einfachen Befehl in der Konsole:
Achtet darauf, dass ihr Groß- und Kleinschreibung beachtet habt, und dass der Name des packages in " " steht (wie ein character).
Wenn ihr Enter gedrückt habt sollte R anfangen loszurödeln, vielelicht gehen auch einige Fenster mit Fortschrittsbalken auf, wenn ihr Windows benutzt.
Das schöne an diesem Befehl ist, dass es auch direkt alle packages installiert, die wir in unserem package benutzen: Sogenannte dependencies. Eine dependency (Abhängigkeit) ist in diesem Kontext ein package, das von einem anderen package gebraucht wird um zu funktionieren. Wir benutzen im package tadaatoolbox zum Beispiel auch die packages dplyr, pixiedust und sjlabelled, deswegen sollte der Befehl diese packages auch gleich mitinstallieren.
Alternativ könnt ihr rechts in RStudio im “Packages”-Tab den “Install”-Button drücken, den namen des packages (tadaatoolbox) eingeben, und dann macht RStudio im Hintergrund genau dasselbe Spielchen mit install.packages().
In diesem Fenster ist auch von Repositories die Rede. Damit ist der Web-Adresse gemeint, von der die packages geladen werden sollen. RStudio sollte da automatisch die schnellste Quelle auswählen, aber wenn ihr mal in die Verlegenheit kommt euch entscheiden zu müssen, versucht am besten folgende Adresse:
https://cloud.r-project.orgUnd da packages auch nur Software sind, und Software auf dem aktuellen Stand gehalten werden will, bietet es sich an sporadisch (spätestens alle paar Monate) mal den Update-Button zu drücken und einfach alles zu aktualisieren, was aktualisiert werden kann.
Das könnt ihr auch aus der Konsole heraus machen indem ihr den Befehl update.packages(ask = FALSE) ausführt. R fragt euch dann, ob ihr sicherheitshalber R neustarten wollt, das könnt ihr tun oder auch nicht, aber wenn ihr es nicht tut, dann solltet ihr auf jeden Fall die R-Session neu starten nachdem ihr alle Updates gemacht habt (RStudio -> Session -> Restart R).
Wieso? Nun ja, packages werden von R geladen, das heißt verfügbar gemacht, und wenn ihr ein package ersetzt (was beim Update passiert), dann zieht ihr damit R praktisch den Boden unter den Füßen weg und es ist sauer weil Dinge anders sind, als sie eben noch waren.
Dieses “verfügbar machen” sieht übrigens so aus:
Wenn ihr diesen Befehl ausgeführt hat, dann läd R für die aktuelle Session das package und ihr könnt die Funktionen darin benutzen.
Normalerweise beginnen eure R-Scripte mit einer Reihe von library()-Befehlen um eure Analyse vorzubereiten und alle benötigten packages zu laden, da ihr diesen Schritt jedes mal wiederholen müsst, wenn ihr eine neue R-Session starten (z.B. beim Neustart von RStudio, Computerneustart etc.).
Außerdem gebt ihr so euren KommilitonInnen eine gute Gelegenheit abzuschätzen, was in eurem Script so passiert, wenn sie direkt erkennen können, welche packages ihr dafür benutzt habt.
Es gibt tausende R-packages, und die meisten davon sind für euch vollkommen uninteressant, aber einige wiederum sind so dermaßen praktisch, dass wir sie uns hier im Detail anschauen.
6.1.1 Quellen
Die wichtigste Quelle für R-packages ist das erwähnte CRAN, kurz für “Comprehensive R Archive Network”. Packages müssen diverse Anforderungen erfüllen, um auf CRAN publiziert zu werden, was eine gewisse Hürde darstellt. Deswegen gibt es diverse packages, die gerade in frühen, potenziell noch nicht ausgereiften Versionen an anderen Stellen verfügbar gemacht werden.
Die wohl populärster dieser Sekundärquellen ist GitHub.
Auf GitHub findet ihr zum Beispiel auch den Quelltext der tadaatoolbox.
Optional könnt ihr packages auch direkt von GitHub installieren, was insbesondere dann interessant ist, wenn das package noch jung und experimentell ist.
In euren normalen Projekten solltet ihr euch nicht auf GitHub-packages verlassen, sondern nach Möglichkeit ausschließlich packages von CRAN benutzen, aber wenn euch nach Abenteuer ist, dann fühlt euch frei:
6.1.2 Maintenance
Packages installieren ist einfach, aber wie jede Software wollen auch R-packages auf dem neusten Stand gehalten werden. Oder zumindest auf einem “nicht total veraltet”-Stand.
Updaten ist ziemlich einfach:
Entweder ihr klickt im “Packages”-Tab von RStudio auf den “Update”-Button, wählt alle Packages aus und installiert die Updates so, oder ihr gebt in der Konsole folgendes ein:
…dann rödelt R die Updates durch. RStudio wird euch an dieser Stelle fragen, ob ihr die R-Session vorher neustarten wollt – das könnt ihr tun, aber er könnt damit auch warten bis ihr alle Updates installiert habt anstatt für jedes einzelne Update die Session neuzustarten.
Wichtig ist auf jeden Fall, dass ihr die R-Session neustartet bevor ihr weiter arbeitet.
Wenn ein package geladen (library()) ist während ihr Updates durchführt, muss die R-Session danach neugestartet werden! Wenn ein package aktualisiert wird während es geladen ist macht das R sehr traurig und Fehler treten auf.
Solltet ihr mal ein packages löschen wollen, aus welchem Grund auch immer, dann geht das entweder auch über den Package-Tab in RStudio (das Kreuzchen rechts neben dem Namen des packages), oder ihr gebt folgendes in der Konsole ein:
…um das package tadaatoolbox zu deinstallieren.
Aber wieso solltet ihr das tun wollen.
Wir haben so viel Arbeit in das package gesteckt.
Wieso nur.
Ihr Monster.
6.2 sjPlot und Co.
sjPlot ist das Aushängeschild der strengejacke-Packages von Daniel Lüdecke, und insgesamt sind seine packages ziemlich ziemlich praktisch für die gängigen sozialwissenschaftlichen Analysen.
Eine umfassende Dokumentation findet ihr unter http://www.strengejacke.de/sjPlot/, und die packages von Interesse installiert ihr wie folgt:
install.packages("sjPlot")
install.packages("sjmisc")
install.packages("sjstats")
install.packages("sjlabelled")6.2.1 sjPlot
Das wichtigste package ist wie erwähnt sjPlot, also laden wir das erstmal:
sjPlot ist ziemlich praktisch wenn ihr mal schnell relativ komplexe Dinge erreichen wollt, in erster Linie Plots und Tabellen.
Die Funktionen im package sind nach ihrem Präfix sortiert:
sjt.für Funktionen, die Tabellen produzierensjp.für Funktionen, die Plots produzieren
Ein paar Beispiele anhand unseres qmsurvey-Datensatzes:
6.2.1.1 Tabellen
Für Kreuztabellen, wie wir sie relativ häufig in QM1 brauchen für unsere nominal- und ordinalskalierten Statistiken:
|
Was für ein Ernährungstyp bist du |
Dein Geschlecht | Total | ||
|---|---|---|---|---|
| keine Angabe | männlich | weiblich | ||
| Andere | 1 | 1 | 5 | 7 |
| Omnivore / Alles | 0 | 11 | 52 | 63 |
| Vegan | 0 | 1 | 5 | 6 |
| Vegetarisch | 0 | 3 | 23 | 26 |
| Total | 1 | 16 | 85 | 102 | χ2=14.201 · df=6 · Cramer’s V=0.264 · Fisher’s p=0.288 |
sjt.xtab(qmsurvey$ernaehrung, qmsurvey$gender,
title = "Kontigenztabelle",
show.cell.prc = TRUE, show.exp = TRUE, show.legend = TRUE)|
Was für ein Ernährungstyp bist du |
Dein Geschlecht | Total | ||
|---|---|---|---|---|
| keine Angabe | männlich | weiblich | ||
| Andere |
1 0 1 % |
1 1 1 % |
5 6 4.9 % |
7 7 6.9 % |
| Omnivore / Alles |
0 1 0 % |
11 10 10.8 % |
52 52 51 % |
63 63 61.8 % |
| Vegan |
0 0 0 % |
1 1 1 % |
5 5 4.9 % |
6 6 5.9 % |
| Vegetarisch |
0 0 0 % |
3 4 2.9 % |
23 22 22.5 % |
26 26 25.4 % |
| Total |
1 1 1 % |
16 16 15.7 % |
85 85 83.3 % |
102 102 100 % |
χ2=14.201 · df=6 · Cramer’s V=0.264 · Fisher’s p=0.291 |
observed values
expected values
% of total
Oder einfache Häufigkeitstabellen:
| val | frq | raw.prc | valid.prc | cum.prc | |
|---|---|---|---|---|---|
| Andere | 7 | 6.86 | 6.86 | 6.86 | |
| Omnivore / Alles | 63 | 61.76 | 61.76 | 68.63 | |
| Vegan | 6 | 5.88 | 5.88 | 74.51 | |
| Vegetarisch | 26 | 25.49 | 25.49 | 100 | |
| NA | 0 | 0 | NA | NA | |
| total N=102 · valid N=102 · x̄=2.50 · σ=0.95 | |||||
Und für lineare Modelle, in deutlich schöner als summary():
model <- lm(zufrieden ~ alter * berufsvorstellung, data = qmsurvey)
tab_model(model, show.std = TRUE)|
Wie zufrieden bist du insgesamt mit dem Psychologiestudium |
|||||
|---|---|---|---|---|---|
| Predictors | Estimates | std. Beta | CI | standardized CI | p |
| (Intercept) | 4.53 | 1.89 – 7.16 | 0.001 | ||
| Dein Alter in Jahren | -0.05 | -0.20 | -0.17 – 0.07 | -0.71 – 0.31 | 0.436 |
|
Ich habe in Bezug auf meine spätere Berufswahl bereits genaue Vorstellungen |
-0.28 | -0.39 | -1.19 – 0.64 | -1.67 – 0.89 | 0.552 |
| alter:berufsvorstellung | 0.01 | 0.46 | -0.03 – 0.06 | -0.94 – 1.87 | 0.521 |
| Observations | 102 | ||||
| R2 / R2 adjusted | 0.007 / -0.023 | ||||
6.2.1.2 Plots
Klassiker: Histogramm mit Normalverteilungskurve:
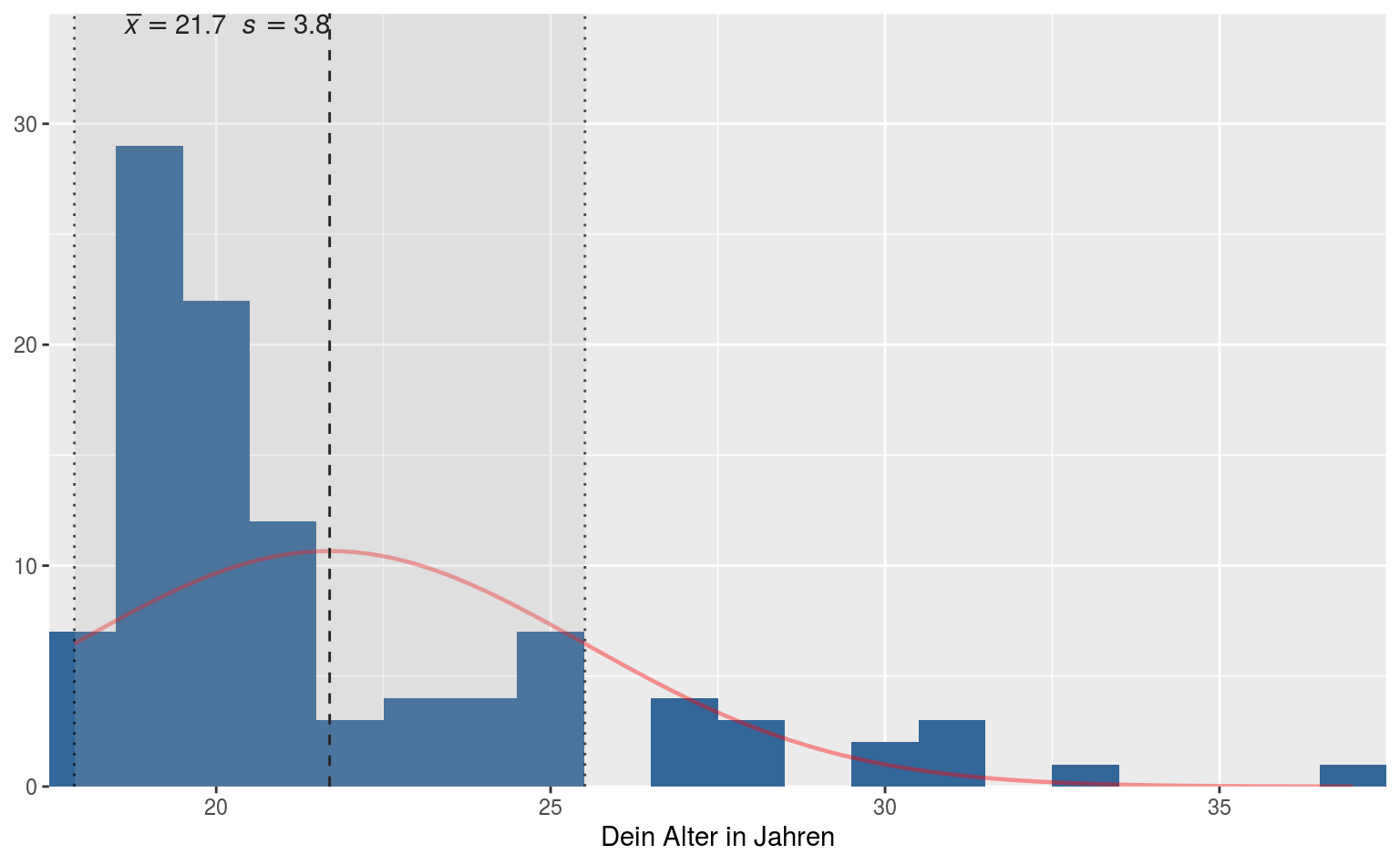
Oder für eine graphische Version einer Kontingenztabelle:
#> Warning in stats::chisq.test(ftab): Chi-squared approximation may be
#> incorrect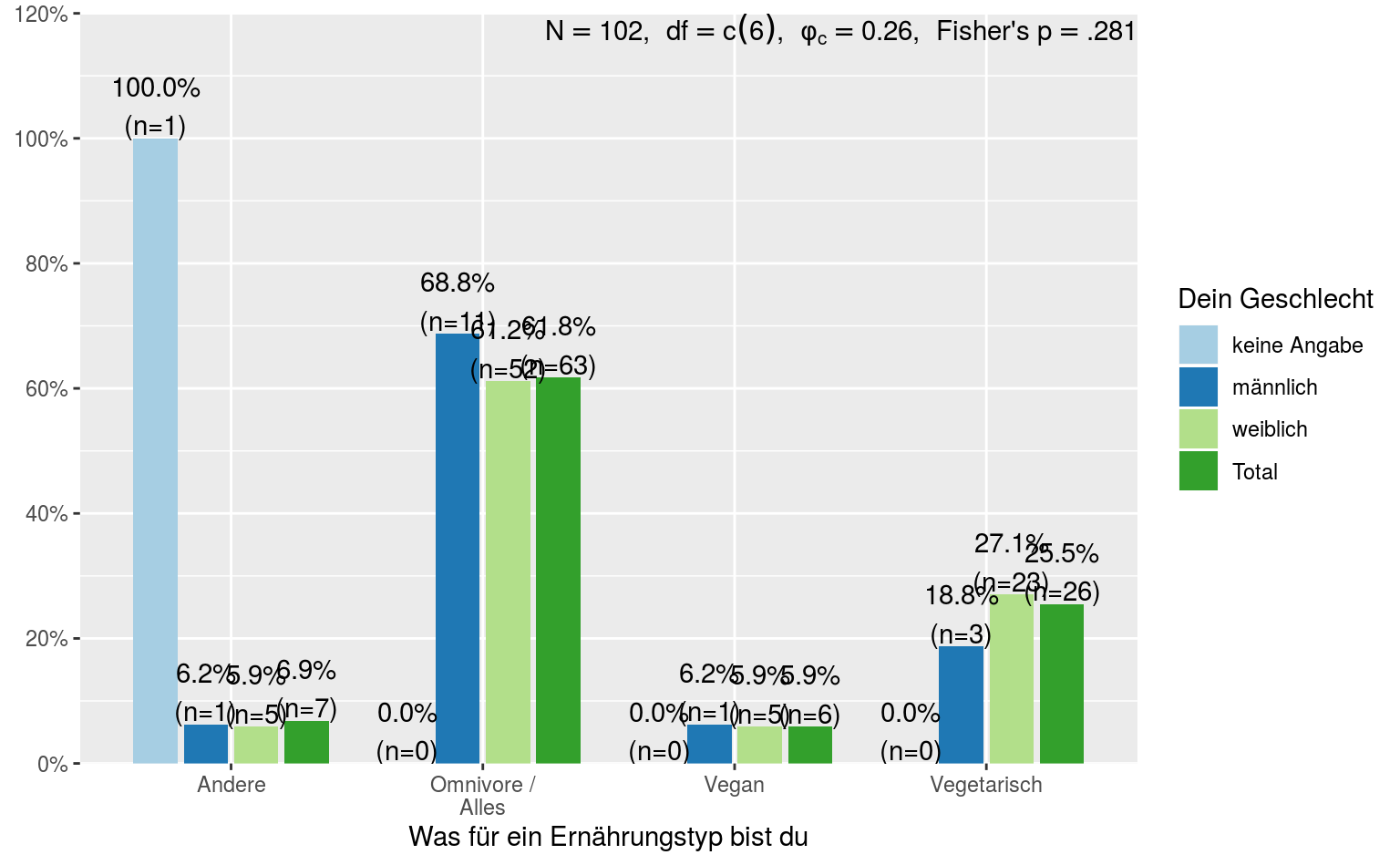
6.2.2 sjmisc
sjmisc ist mehr so ein package für Datenbearbeitung, deshalb fällt es mir schwer da demonstrativ eine Funktion rauszuziehen. Im Fokus liegt unter Anderem variable recoding, das heißt falls ihr mal eine Variable klassieren müsst, könnte das mit sjmisc ganz dankbar werden.
Wenn wir alter in 3 Klassen umwandeln wollen, sähe das also zum Beispiel so aus:
#> [1] 1 2 1 1 1 2 1 1 1 2 1 1 1 1 2 1 1 1 1 1 1 2 1 1 1 3 2 3 1 1 1 1 1 1 2
#> [36] 1 3 1 2 1 1 2 1 1 1 2 1 1 1 1 1 1 2 2 1 1 1 1 1 1 1 2 1 2 3 1 1 1 1 2
#> [71] 1 1 2 3 1 1 1 1 2 1 1 1 1 1 2 2 1 1 1 3 2 1 1 2 1 1 3 1 1 1 2 1
#> attr(,"label")
#> alter
#> "Dein Alter in Jahren"Um die rekodierte Variable dann in unserem Datensatz zu speichern sähe das dann mit Hilfe von dplyr so aus:
6.2.3 sjlabelled
Wenn ihr Daten aus SPSS einlest (oder ihr auf labelled data steht), dann haben eure Datensätze vermutlich labels. In base R ist das generell nicht so gängig, aber seit einiger Zeit sind die Tools dafür recht populär geworden. Labels sind zum Beispiel dafür zuständig, dass in eurer Variablenansicht jede Spalte eine Beschreibung hat, was im Falle der sjt.*-Funktionen auch dafür sorgt, dass eure Tabellen sauber beschriftet sind.
Ihr müsst sjlabelled in der Regel nicht explizit laden, sjPlot benutzt das unter der Haube sowieso, aber wenn ihr eure Labels mal ändern wollte oder ihr einen neuen Datensatz zusammensteckt, wo ihr eure Variablen labeln wollt, dann ist das package euer Freund.
Die Funktionen dafür sind folgende:
set_label: Für Variablenlabelsset_labels: Füe Value labels, sprich einzelne Merkmalsausprägung (1= “niedrig” etc.)get_label: Variablenlabels anzeigenget_labels: Value labels anzeigen
6.3 Das tidyverse Hail Hadley!
Das tidyverse ist eine Ansammlung von packages, die alle mehr oder weniger gut miteinander auskommen und auf ähnliche Art zu benutzen sind.
Eine Übersicht und ganz viel Dokumentation und Beispiele findet ihr auf http://tidyverse.org.
Es gibt ein catchall R-package, das die wichtigstens packages für euch installiert:
Ihr könnt danach entweder das package laden und habt damit die wichtigsten Funktionen parat, oder ihr ladet die packages nach Bedarf einzeln.
Ich würde zu letzterem raten, weil ihr so eher ein Gefühl dafür bekommt welches package für welche Funktionen zuständig ist, und nebenbei geht es auch ein bisschen schneller.
Hier eine kurze Übersicht über die wichtigsten tidyverse-packages/Funktionen für alltägliche Aufgaben:
%>%: Der pipe-Operator aus magrittr, der von den meisten der packages re-exportiert wird- ggplot2: Für Visualisierungen
- dplyr: Datenmanipulation
mutate: Neue Variablen erstellen/ bestehende Verändernselect: Variablen auswählenfilter: Datensätze filterngroup_by: Gruppieren…summarize: Und zusammenfassen
- tidyr: Datenmanipulation
gather: Konvertiert von wide in long format (mehrere Spalten zu zwei zusammenfassen)spread: Gegenstück, konvertiert von long in wide format
6.4 Die tadaatoolbox
Die tadaatoolbox ist das Produkt von Lukas’ und Tobis langjähriger Frustration mit diversem Kleinkram in R, wie etwa der Optik von statistischen Testoutput.
Im Fokus steht in erster Linie schnelles, einfaches Output mit möglichst wenig Aufwand. Zusätzlich bietet die Toolbox einige Sammelfunktionen um beispielsweise diverse nominal- oder ordinalskalierte Statistiken auf einen Rutsch anzuzeigen, optimiert auf Output in RMarkdown-Dokumenten (Siehe Berichte).
Eine Übersicht gibt es auf der Seite der Toolbox: http://tadaatoolbox.tadaa-data.de
Beispielhaftes Testoutput findet sich dort, und eine Funktionsreferenz gibt’s da auch.
6.5 Addendum: “Installier mal alles”
Hier ein Stück Code, das ihr im Zweifelsfall einfach copypasten könnt.
Es sollte euch so ziemlich alles oder zumindest das meiste an (vorerst) relevanten packages installieren, und dient mehr so der Vollständigkeit.
install.packages("magrittr")
install.packages("ggplot2")
install.packages("dplyr")
install.packages("tidyr")
install.packages("stringr")
install.packages("devtools")
install.packages("forcats")
install.packages("readr")
install.packages("lubridate")
install.packages("purrr")
install.packages("readxl")
install.packages("haven")
install.packages("rvest")
install.packages("scales")
install.packages("sjmisc")
install.packages("sjstats")
install.packages("sjlabelled")
install.packages("sjPlot")
install.packages("tadaatoolbox")
install.packages("cowplot")
install.packages("waffle")
install.packages("ggrepel")
install.packages("psych")
install.packages("DescTools")
install.packages("ggthemes")
install.packages("hrbrthemes")
install.packages("rmarkdown")
install.packages("knitr")
install.packages("mosaic")
install.packages("htmlwidgets")
install.packages("DT")
install.packages("viridis")
install.packages("RColorBrewer")
install.packages("nortest")
install.packages("pixiedust")
install.packages("plotly")
install.packages("revealjs")
install.packages("rpivotTable")
install.packages("artyfarty")
install.packages("car")
install.packages("colourpicker")
install.packages("dygraphs")
install.packages("lsr")
install.packages("vcd")
install.packages("ryouready")
install.packages("esc")
install.packages("ez")
install.packages("afex")Die Ruby-Leute und ihre Gems seien mal dahingestellt↩